por Tayná Fiúza

Fonte: https://brasil.mongabay.com/2016/05/cientistas-fazem-dolorosa-descoberta-um-sapo-capaz-injetar-veneno/ Créditos: Carlos Jared
A perda de biodiversidade ao redor do mundo preocupa e ameaça o tênue equilíbrio que permite que parte da população humana se mantenha em condições dignas de vida. Desde 1970, o Living Planet Index é utilizado para estimar as condições de biodiversidade ao redor do mundo a partir da abundância de populações não-humanas. O número, que começou em 1, hoje se encontra em 0.68 e preocupa pesquisadores. Do ponto de vista estritamente técnico, muito conhecimento e potenciais aplicações são perdidas quando perdemos biomas.
Na contramão de outras nações que fazem grandes esforços para catalogar e preservar seu patrimônio genético, no Brasil observamos o maior volume de queimadas já registrado no Pantanal e um aumento de mais de 30% na incidência de desmatamentos na Amazônia. Além disso, biomas menos catalogados como a Caatinga podem sofrer perdas de diversidade ainda maiores, e mais silenciosas. Um dos métodos de catalogação de espécies que estimula a preservação, avança o conhecimento científico e abre portas para a aplicação com retorno econômico é o sequenciamento.
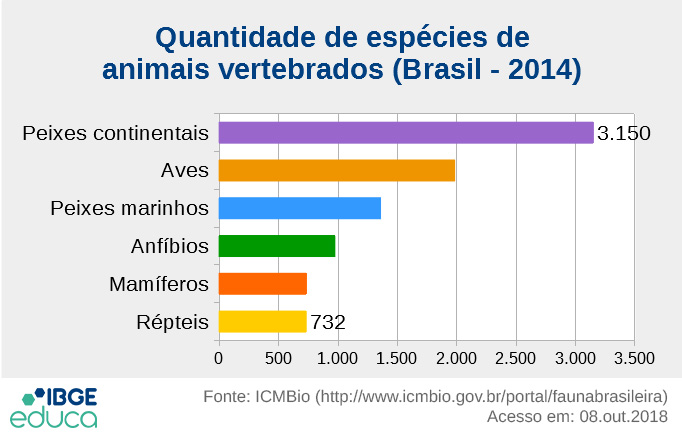
No Brasil, são mais de 8900 espécies de vertebrados e 120 mil de invertebrados.
Fonte: https://educa.ibge.gov.br/jovens/conheca-o-brasil/territorio/18309-a-fauna-brasileira.html
Já introduzimos a importância de conhecermos nossas espécies aqui, seja visando uma aplicação biotecnológica específica e prospectando diferentes genes, seja para entender a história evolutiva de nossa biodiversidade como fim em si mesmo. Para ilustrar as possibilidades, trago a história de duas espécies que são as últimas de seus respectivos grupos: as tuataras (Sphenodon punctatus), da Nova Zelândia e os peixes Bowfin (Amia calva) , do leste da América do Norte.
Identificado primeiro vertebrado com dois genomas mitocondriais
As tuataras, répteis endêmicos da Nova Zelândia, foram objeto de uma descoberta singular: seu material genético abriga dois genomas de mitocôndrias, organelas de fonte de energia para as células. Esse fenômeno não é particular às tuataras, mas a espécie foi a primeira dentre os vertebrados a possuir tal característica determinada.
Além do DNA nuclear, os animais possuem um genoma extra-nuclear nas mitocôndrias e ao examinar esses diferentes materiais podemos identificar eventos evolutivos e entender a história das diferentes espécies. Nesse caso, as tuataras são as últimas representantes vivas de sua ordem (Rhynchocephalia) e a evidência de dois genomas mitocondriais abre caminho para muitas novas perguntas.
Por KeresH — Trabalho Pessoal, CC BY-SA 3.0, https://commons.wikimedia.org/w/index.php?curid=4538993
Anteriormente, cientistas buscavam entender a ausência de alguns genes mitocondriais padrão. Foi apenas com um trabalho de genômica comparativa cruzando o DNA de tuataras contra o de outros répteis que as cientistas Ella Buring e Vanessa González puderam observar uma grande quantidade de sequências extras não reportadas previamente.
Foi então que, com dados de um novo sequenciamento de genoma completo, as pesquisadoras identificaram e montaram o segundo genoma mitocondrial, contendo cerca de 10% de diferença em relação ao DNA mitocondrial padrão. Se a observação que foi feita apenas com um espécime for confirmada em outros indivíduos, cientistas terão mais uma peça para montar a história da espécie e de sua ordem dentro dos répteis.
Peixe bowfin surpreende pesquisadores com desenvolvimento alternativo de barbatana
Já os bowfins, peixes do leste da América do Norte, são os últimos sobreviventes do grupo Halecomorphi. Essa espécie já intrigava pesquisadores devido suas características mistas: algumas típicas de peixes ancestrais, como respiração com aparatos semelhantes à da respiração pulmonar; e outras de grupos mais derivados como uma cauda reduzida e escamas simples.
Já de posse do sequenciamento do genoma do animal, o então estudante de doutorado Brent Hawkins decidiu analisar o transcriptoma do Amia calva ao longo do desenvolvimento de suas nadadeiras e também fez uso de hibridização in situ para observar a localização dos genes ativados ao longo do desenvolvimento.
Por Stan Shebs, CC BY-SA 3.0, https://commons.wikimedia.org/w/index.php?curid=2699630
Para sua surpresa, vários genes anteriormente considerados como críticos para o crescimento das nadadeiras não estavam presentes, como o fator de crescimento fibroblástico 8 (Fgf8). A capacidade da espécie de se desenvolver sem um gene tão essencial quanto o Fgf8 aponta uma flexibilidade na reprogramação celular para o desenvolvimento do corpo destes peixes.
Outras descobertas envolvem a expressão alta de um RNA o qual não é traduzido em proteína - essa sequência pertence ao HoxD14, bastante encontrado em peixes mais ancestrais na árvore filogenética. Nos bowfins, a sequência do HoxD14 não codifica uma proteína funcional, e portanto o trabalho de entender a ação do RNA de HoxD14 na regulação de genes Hox nessa espécie ainda está em aberto.
Esses dados ressaltam a importância de lembrar que espécies com características morfológicas antigas que hoje habitam o planeta não podem ser consideradas como se fossem ancestrais das atuais, uma vez que se desenvolveram de um ancestral comum do mesmo modo que as demais espécies.
Há um número crescente de informações curiosas e de importância biotecnológica que são obtidas através de sequenciamentos de DNA ou RNA e as possibilidades são tantas quanto a seleção natural permitiu que chegassem até aqui. Ao longo dos próximos anos ainda encontraremos muitos “primeiros” e, naturalmente, os esquemas que explicam a história da vida serão revisados e atualizados de acordo. Cientistas em um país com tantas espécies endêmicas como o Brasil poderiam estar em vantagem nessa aventura, com a ajuda de agências de financiamento. Até lá, vamos acompanhar as descobertas instigantes sobre a fauna de outras nações.
Glossário:
- Endêmico: restrito a determinada região geográfica (diz-se de espécie, organismo ou população).
- Ordem: é um taxon de alto nível hierárquico utilizado no sistema de classificação científica dos seres vivos para agrupar famílias constituídas por espécies que apresentam entre si um elevado grau de semelhança morfofuncional.
- Transcriptoma: conjunto completo de transcritos (RNAs mensageiros, RNAs ribossômicos, RNAs transportadores e os microRNAs) de um dado organismo, órgão, tecido ou linhagem celular.
- Hibridização in situ : método que permite identificar o locus cromossômico onde se localiza uma determinada sequência de DNA previamente clonada.
Você pode gostar de ler:
Quando teremos os genomas da nossa biodiversidade?
https://bioinfo.imd.ufrn.br/transcricaoemdia/opiniao/eg-002
Para saber mais:
Perda de biodiversidade ameaça desenvolvimento científico e inovação, alertam especialistas
WWF (2020) Living Planet Report 2020 - Bending the curve of biodiversity loss. Almond, R.E.A., Grooten M. and Petersen, T. (Eds). WWF, Gland, Switzerland
https://f.hubspotusercontent20.net/hubfs/4783129/LPR/PDFs/ENGLISH-SUMMARY.pdf
Intern Helps Find First Vertebrate With Two “Powerhouse” Genomes | Smithsonian Voices | National Museum of Natural History Smithsonian Magazine
Evidence of two deeply divergent co-existing mitochondrial genomes in the Tuatara reveals an extremely complex genomic organization | Communications Biology

