por Tayná Fiuza
Em novembro de 2020, foram anunciados os resultados da CASP, Avaliação Crítica de Predição de Estruturas (Critical Assessment of Structure Prediction, em inglês) e o programa AlphaFold, desenvolvido pela empresa DeepMind, conquistou o primeiro lugar. Os organizadores da competição mencionaram o progresso sem precedentes na capacidade de predição de estruturas obtida pelo método computacional proposto. O valor de 87/100 obtido no teste de distância global, que compara a estrutura predita com a estrutura determinada experimentalmente, é um salto em relação aos 68.5/100 obtidos pela primeira versão do AlphaFold, que venceu a edição anterior da CASP em 2018. A equipe por trás do AlphaFold participou da competição com uma solução de predição ab initio de estruturas proteicas, isto é, uma predição de modelos de estrutura terciária a partir apenas da sequência de aminoácidos disponibilizadas, sem modelos pré-validados.
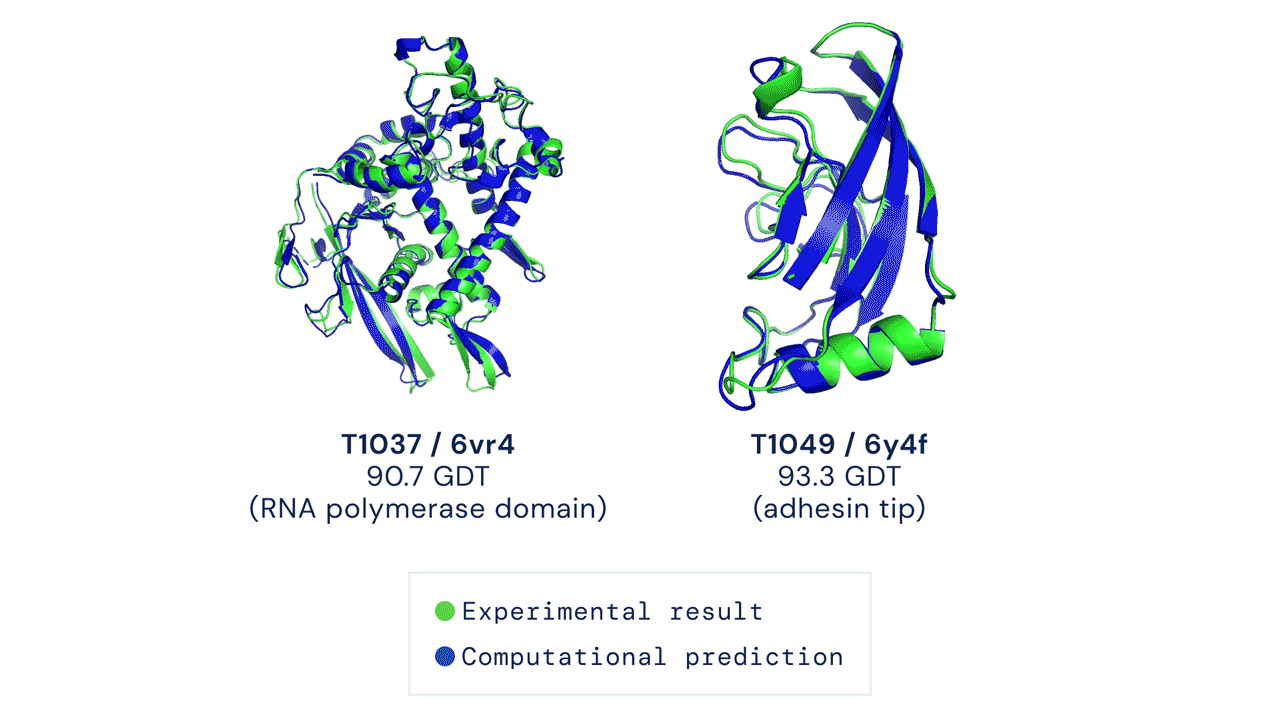 Legenda: Exemplos de sobreposição entre a estrutura predita pelo AlphaFold (azul) e a estrutura determinada experimentalmente, modelo ouro para comparação (verde). Em ambos os casos, a medida de similaridade de estruturas GDT está acima de 90, marco de equivalência entre duas estruturas. Fonte: https://deepmind.com/blog/article/alphafold-a-solution-to-a-50-year-old-grand-challenge-in-biology
Legenda: Exemplos de sobreposição entre a estrutura predita pelo AlphaFold (azul) e a estrutura determinada experimentalmente, modelo ouro para comparação (verde). Em ambos os casos, a medida de similaridade de estruturas GDT está acima de 90, marco de equivalência entre duas estruturas. Fonte: https://deepmind.com/blog/article/alphafold-a-solution-to-a-50-year-old-grand-challenge-in-biology
No algoritmo de aprendizagem de máquina AlphaFold, o conjunto de treinamento foi composto de cerca de 170.000 estruturas proteicas disponíveis no PDB, Banco de Dados de Proteínas (Protein Data Bank, em inglês). As sequências utilizadas como entrada do algoritmo de Aprendizagem Neuronal Profunda foram empregadas para a predição da distância entre pares de aminoácidos e dos ângulos entre as ligações que conectam os aminoácidos, que compõem as métricas necessárias para a determinação da estrutura tridimensional da proteína. Uma outra maneira de entender a estrutura proteica é através de grafos espaciais, em que arestas conectam aminoácidos (nós) próximos. Os dados de treinamento são usados para construir tais grafos e posteriormente, informações de sequências relacionadas evolutivamente, alinhamentos de múltiplas sequências e representações dos pares de aminoácidos são empregadas para melhorar o grafo produzido.
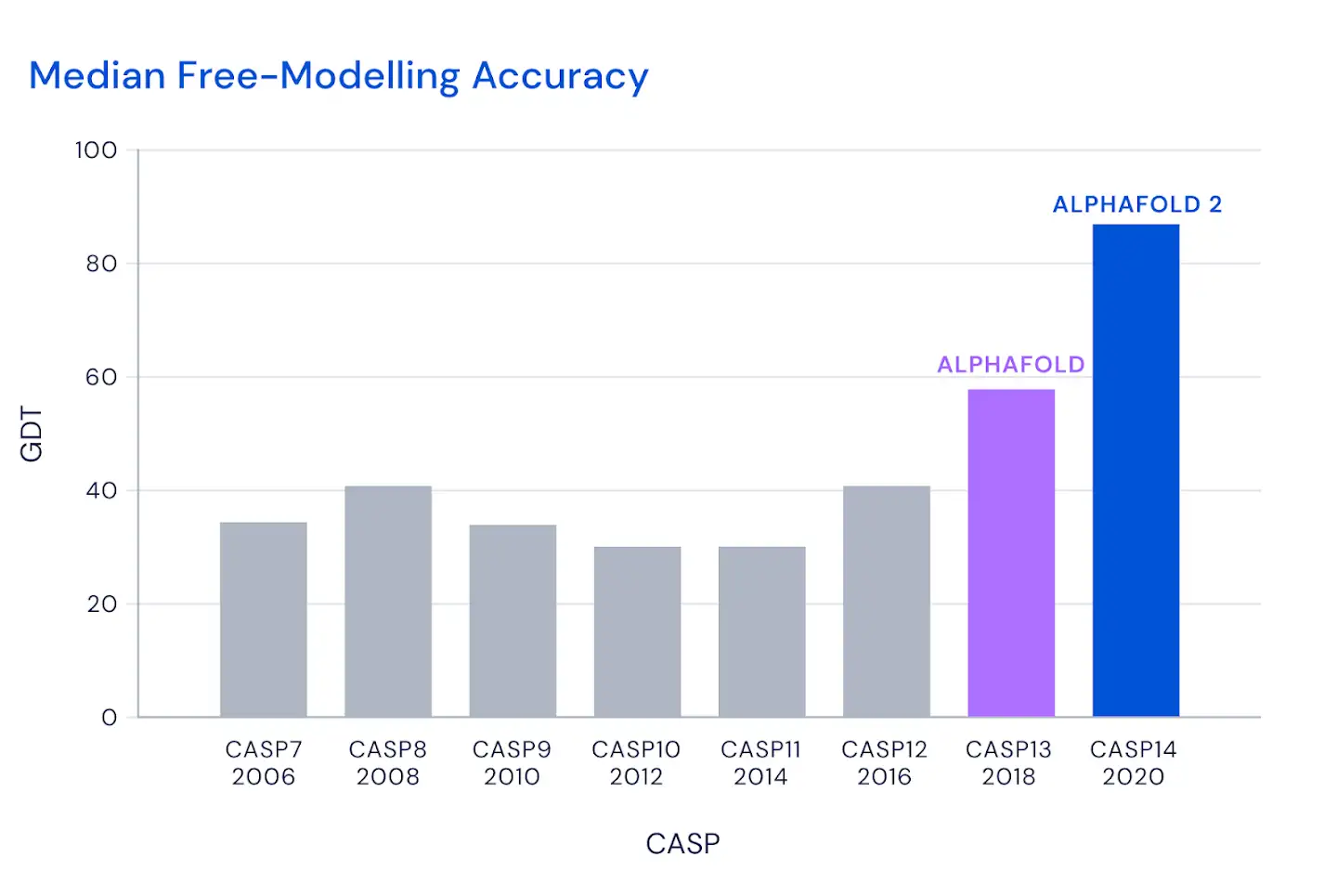 Legenda: Acurácia dos melhores algoritmos do ano para a categoria Ab-Initio (Free Modelling). Fonte: https://deepmind.com/blog/article/alphafold-a-solution-to-a-50-year-old-grand-challenge-in-biology
Legenda: Acurácia dos melhores algoritmos do ano para a categoria Ab-Initio (Free Modelling). Fonte: https://deepmind.com/blog/article/alphafold-a-solution-to-a-50-year-old-grand-challenge-in-biology
Na CASP, os grupos são convidados a predizer a estrutura de diversas entidades proteicas e o desempenho do AlphaFold não foi equiparável em todos os casos. Para algumas proteínas, de acordo com os avaliadores, não ficou claro se as divergências eram fruto de erro de predição ou de artefato gerado no experimento de determinação e geração de modelo. Já para outras, especialmente aquelas cujos modelos estão em complexos proteicos ou são provenientes de Ressonância Magnética Nuclear (NMR) há uma considerável taxa de erro. Especialistas atribuem essas diferenças a mudanças que ocorrem na estrutura proteica pelas interações inerentes a formação de complexos e a métodos de conversão dos dados brutos de NMR para os modelos disponíveis.
Alguns cientistas se mostram bastante otimistas quanto aos resultados e impactos do algoritmo desenvolvido e disponibilizado, apontando uma nova era na identificação das funções de proteínas e de suas variantes implicadas em doenças. Outros indicam vantagens não apenas na área da saúde, mas também no desenvolvimento de enzimas mais eficazes e de maior aplicabilidade no contexto de biorremediação e bioconversão de poluentes como o plástico.
Outro grupo se mostra mais cauteloso em suas declarações, reconhecendo a importância do trabalho desenvolvido ao passo que ressalta algumas limitações do método e do campo de estudo como um todo. Como em diversos algoritmos de aprendizado de máquina, a predição desenvolvida pelo AlphaFold por si não traz insights biologicamente ou quimicamente compreensíveis sobre como os dados de entrada foram combinados e geraram os resultados observados.
Além disso, alguns apontam que o tamanho do conjunto de treinamento corresponde a cerca de 5% do proteoma humano total e que as amostras experimentalmente disponíveis atualmente são, em sua maior parte, as de mais fácil obtenção experimental. Alguns últimos criticam a noção de que predizer estruturas implica em compreender a função desta estrutura, uma vez que proteínas similares podem apresentar funções distintas e proteínas distantes em sua sequência e estrutura podem desempenhar papéis análogos.
Interpretações à parte, o que se viu foi que metade dos competidores da CASP este ano desenvolveram estratégias relacionadas a aprendizagem de máquina profunda, demonstrando mais uma vez sua grande capacidade de resolver problemas biológicos complexos. Iterações e melhorias já foram feitas no método AlphaFold de 2018, e se espera que o mesmo seja feito para segunda versão (2020). Sua implementação com código aberto permitirá que um grupo maior de cientistas avancem suas pesquisas na área, seja avaliando os resultados obtidos, seja construindo seus próprios métodos, ou mesmo utilizando-o em estudos aplicados que dependam de predições da estrutura tridimensional de proteínas.
Glossário:
Cristalografia de raio X: Raios X são aplicados em uma amostra cristalizada e a difração destes raios em diferentes direções e ângulos forma padrões. Estes padrões podem ser estudados para calcular a densidade eletrônica da amostra original. A partir dessas nuvens de densidade se pode inferir a posição de átomos, presença de ligações químicas e outras informações.
Estrutura terciária: Estrutura tridimensional global de única cadeia proteica. Os demais níveis de organização proteica se referem a sequência (primária), arranjos locais ordenados (secundária) e estrutura global com duas ou mais cadeias proteicas (quaternária).
Aprendizagem Neuronal Profunda: Acontece em redes neuronais profundas, ou seja, aquelas em que há um grande número de camadas entre o ponto de entrada dos dados e a predição calculada. Em cada camada diferentes características recebidas da camada anterior são combinados em funções matemáticas e os resultados são passados para a camada posterior.
Ressonância Magnética Nuclear: Fenômeno físico em que núcleos atômicos expostos a um campo magnético constante e forte são perturbados por um campo magnético oscilante e fraco. Um sinal dessa perturbação é produzido, tendo uma frequência característica para tal núcleo. Nem todos os núcleos atômicos presentes numa amostra irão gerar este sinal, o que permite obter informações das estruturas em tal amostra.
Para Saber Mais:
Behind the screens of AlphaFold | Opinion | Chemistry World
https://www.chemistryworld.com/opinion/behind-the-screens-of-alphafold/4012867.article
AlphaFold: a solution to a 50-year-old grand challenge in biology | DeepMind
https://deepmind.com/blog/article/alphafold-a-solution-to-a-50-year-old-grand-challenge-in-biology
‘It will change everything’: DeepMind’s AI makes gigantic leap in solving protein structures
https://www.nature.com/articles/d41586-020-03348-4
AlphaFold: Using AI for scientific discovery | DeepMind
https://deepmind.com/blog/article/AlphaFold-Using-AI-for-scientific-discovery