por Epitácio Farias Filho, Ivan Álisson de Lima e Ruth Setúbal

Imagem: Mikhail Konoplev
O que é e por quê devo aprender?
RDD significa conjuntos de dados distribuídos resilientes. Em outras palavras, quando você transforma um dado em uma RDD, estes dados são fatiados em várias porções menores e distribuídos, podendo ser armazenados na memória de diversas máquinas de um cluster (Conjuntos de máquinas que trabalham juntas), para então serem operados em paralelo, como é ilustrado na imagem 1.
Programar com Spark RDDs tem muitas vantagens, são algumas:
Tolerância a falhas: Em caso de falha em um nó, o Spark solicita a outro nó que re-calcule o processo perdido.
Paralelização das atividades: Processamento simultâneo em diversas máquinas.
Divisão/partição das atividades: Distribuição dos dados para várias máquinas.
Processamento in-memory: Processamento executado na memória RAM do sistema.
“Call-by-need”: Calcula uma operação somente quando seu resultado é requisitado.
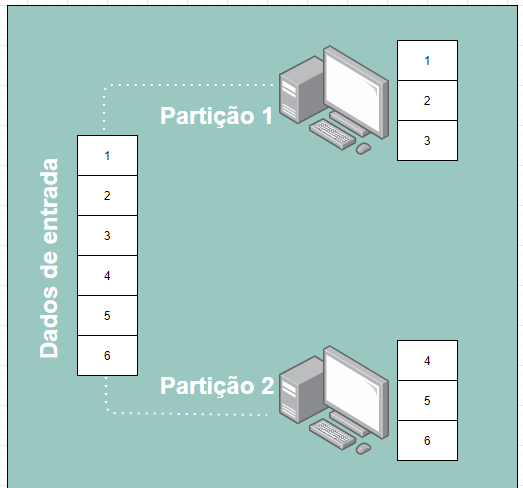
Imagem 1. Representação de como o RDD particiona os dados. Fonte: Própria
Trabalhando com RDDs
Uma das principais características dos RDDs é serem imutáveis, ou seja, uma vez criados não é possível alterá-los. A alternativa para isso é a criação de novos RDDs, que podem ser feitos através de dois tipos de operações: Transformação e Ação.
Na transformação são aceitas coleções já existentes como entrada que irão retornar em novos RDDs, enquanto na ação é retornado um resultado final do cálculo para o driver, através do carregamento de um conjunto de dados em um armazenamento externo de qualquer fonte suportada pelo Hadoop (ex: csv, txt, JSON, e etc). Logo, um RDD de entrada não é alterado, mas sim transformado por meio de operações em um novo ou novos RDDs. Alguns exemplos de operações de Transformação e Ação.
Uma vez que estas operações foram compreendidas é possível escolher uma das formas de criação de RDDs para se dar início.
Criando RDDs
Existem algumas formas de se criar RDDs:
- Paralelização: Geralmente, quando se está aprendendo a utilizar o Spark, o mais comum é começar utilizando o método de paralelização, que consiste em pegar uma coleção de RDDs já existente e submetê-la ao Spark Context parar criar diversas partições. Estas partições podem ser criadas automaticamente ou de forma manual utilizando por exemplo: e.g. sc.parallelize(data, 9) em que o número dado de partições foi 9. Após isso, o próprio Spark executa uma tarefa para cada uma das partições que são enviadas aos servidores de processamento, mais conhecidos como “nós”, onde são executadas as reais aplicações.
- Conjuntos de dados: O Spark também permite a criação de RDDs a partir de um conjunto de dados externos, que podem ser de arquivos de texto, arquivos de sequências e qualquer outra fonte de dados externos suportada pelo Hadoop. Para desenvolvimento de um arquivo de texto é indicado utilizar o método de TextFile disponível no SparkContext. Através de um endereço de URL que pode ser de uma máquina local ou do Hadoop Files System (HDFS) o arquivo é então lido em forma de coleção de linhas. É de suma importância para se ter êxito no processo que o arquivo local e os nós de trabalho compartilhem o mesmo destino/caminho do sistema de arquivos local. Para isso acontecer se sugere utilizar um Sistema de Arquivos de Redes (NFS) em que é possível compartilhar os arquivos entre máquinas de uma rede como se estivessem em um mesmo disco rígido. Outra solução é copiar o arquivo para os nós de trabalho.
O Spark ainda oferece suporte para diversas formas de leitura para arquivos de texto, como por exemplo:
Formas para se ler a partir de RDDs
sparkContext.textFile()
sparkContext.wholeTextFiles()
Formas para se ler a partir do arquivo local ou por HDFS
spark.read.text()
spark.read.textFile()
Quais são suas aplicações na Bioinformática?
Com o advento do sequenciamento da nova geração e o rápido desenvolvimento de tecnologias para ele, o crescimento de dados biológicos e as ferramentas da bioinformática não processavam em tempo hábil essa quantidade de dados, então se fez necessário a aplicação de uma tecnologia de computação distribuída para que o processamento desses dados pudessem ser realizados. Os pesquisadores viram que no ramo da tecnologia da informação já existia alguns frameworks que trabalhavam com essa funcionalidade, como por exemplo o Hadoop e o Apache Spark, comparando esses frameworks foi visto que o Apache Spark tem uma melhor funcionalidade, pois eles inserem a tecnologia dos RDDs, apresentam uma melhor performance em relação a velocidade de processamento e também pela usabilidade dele a partir de Interface de Programação de Aplicativos (APIs), e sua compatibilidade com R, Python, Java e Scala.
Colocando a mão na massa!
Agora vamos seguir um exemplo de implementação para que possamos observar como é simples utilizar o Spark. Além disso faremos uma breve comparação entre o tempo de execução necessário utilizando métodos mais tradicionais e o Spark.
Caso: Para esse exemplo imagine que em um dado momento você precisa contabilizar o número de mudanças de bases presentes em um conjunto de sequências nucleotídicas ou de DNA, para que possa então identificar possíveis SNPs (Single nucleotide polymorphisms). Nesse contexto, a criação de uma lógica que execute essa função é bem simples, porém essa mera atividade pode tornar-se impraticável à medida que o número de sequências ou seu tamanho aumenta. Por essa razão a aplicação de ferramentas que façam uso de paralelismo, como o Spark, são muito bem vindas.
Entendendo a lógica da tarefa:
Contabilizar SNPs é uma tarefa simples, dada duas ou mais sequências alinhadas basta tratarmos essas sequências como uma matriz, onde cada sequência é uma linha e cada letra é uma coluna.
Ex.:
Col - 1 2 3 4 5 6 7 8 9 10 11 12 13 14 14 16 17 18
Seq 1 - A A G A T G A T G G A T G A T G A T
Seq 2 - A A G A C A A T C G A T G A T G A T
⋮
Seq N - A A G A C A A T C G A C G A T G A T
SNPs * * * *
No exemplo acima identificamos, visualmente, as colunas 5, 6, 9 e 12, representadas pelo sinal de asterisco (*), como possíveis posições com mudanças de base. De maneira análoga executaremos esse processo em um arquivos fasta contendo diversas sequências a serem exploradas (um arquivo fasta que pode ser utilizado neste script pode ser baixado aqui), o código em python para essa tarefa é o seguinte:
1 - Iniciamos com o carregamento das bibliotecas e dos arquivos
# Imports
import numpy as np # Lida com arrays
import random # Gera valores aleatórios
import matplotlib.pyplot as plt # Gera os gráficos
from tqdm import tqdm # Barra de progresso para o código
# Bibliotecas para utilizar o Spark
import findspark # Encontra o Spark
from pyspark.sql import SparkSession # Inicia a sessão do Spark
findspark.init()
2 - Preparação dos dados carregados
# Load Fasta
# Local do arquivo
path = 'X:\Local_Do_Arquivo\fasta.afa'
# Abre o arquivo e lê
raw = open(path).read()
# Separa as amostras do arquivo usando o '>'
# como identificador de início de sequência
file = raw.split('>')
# O split gera elementos vazios na lista,
# por isso precisamos removê-los
# Remove vazios
file = [ x for x in file if x]
# Agora separamos cada sequência do seu identificador,
# utilizando o '\n' como guia
file = np.array([x.split('\n', 1) for x in file])
# O resultado dos passos anteriores gera listas dentro de
# listas, por isso precisamos remove as listas aninhadas
file = file.flatten()
# Agora removemos as quebras de linhas de dentro
# das sequências
file = [x.replace('\n', '') for x in file]
# Como o resultado é uma lista contendo o cabeçalho da
# seguido da sequência de maneira intercalada
# utilizamos isso para criar listas separadas de
# cabeçalhos e sequências
seqList = np.array([ file[idx] for idx in range(len(file)) if idx%2!=0])
3 - Para executarmos nossa busca precisamos que as sequências sejam armazenadas em uma matriz, como comentamos anteriormente.
# Transformando a string da seqência em uma lista
seqList = [list(x) for x in seqList]
# Removendo sequências com tamanho errado
tam = max(len(l) for l in seqList)
seqList = [ sub for sub in seqList if len(sub) == tam]
# Transformando em matriz
seqList = np.array(seqList)
4 - Após este passo podemos iniciar a contagem, primeiro demonstraremos o tempo de execução usando uma abordagem convencional.
# Contagem usando CPU
AMNO = list('ARNDCQEGHILKMFPOSUTWYVBZXJ')
NUCL = list('ATCG')
def countPos(mtrx):
"""
Conta o número de mutações
"""
columns = mtrx.shape[0]
resp = []
for ii in range(columns):
resp.append(np.char.count("".join(mtrx[:,ii]), AMNO))
return resp
# Marca o início do processo
start = timeit.default_timer()
# Conta as mutações
finalCPU = countPos(seqList)
# Marca o fim do processo
stop = timeit.default_timer()
# Mostra o resultado
print(f'CPU\nTempo de execução: {stop - start:.5f}s')
CPU
Tempo de execução: 0.21005s
5 - Agora podemos verificar o que acontece ao passarmos o nosso processo para o Spark
# Marca o inicio do processo
start = timeit.default_timer()
# Inicializar o Spark
spark:SparkSession = SparkSession.builder \
.master("local[*]") \
.appName("Conta_Mutacoes") \
.getOrCreate()
# 1 - np.transpose é utilizado para inverter as posições das
# colunas e das linhas
# 2 - parallelize distribui os processos entre os recursos
# disponíveis, nesse exemplo núcleos do processador
# 3 - map, aplica uma função para cada subdivisão passada pelo RDD
# 4 - Lambda, recebe a função que faz a contagem dos caracteres
rdd = spark.sparkContext.parallelize(np.transpose(seqList))\
.map(lambda x: np.char.count("".join(x), AMNO))
# Armazena o resultado gerado pelo Spark
finalSpark = rdd.collect()
# Finaliza o processod o Spark
spark.stop()
# Marca o fim do processo
stop = timeit.default_timer()
# Mostra o resultado
print(f'Spark\nTempo de execução: {stop - start:.5f}s')
Spark
Tempo de execução: 3.68239s
Imagino que nesse momento você esteja se perguntando: ‘Ué na abordagem tradicional o processamento de 900 sequências durou apenas 210 ms, contra 3.70 s no Spark, isso quer dizer que o Spark é pior?’
Excelente observação, como vimos o Spark se saiu ‘pior’ que o método ‘tradicional’, mas isso tem uma explicação simples: a primeira abordagem começa a processar os dados assim que mandamos, porém o Spark tem uma etapa a mais, ele precisa instanciar o processo, pegar os dados, separar utilizando o RDD e só após isso o cálculo será executado.
Portanto, se o tamanho dos dados inseridos forem pequenos demais, o Spark sempre levará aproximadamente 4 segundos, no mínimo. Logo, a utilização dessa abordagem só se mostra interessante se o conjunto de entrada for suficientemente grande, para demonstrar esse ponto, o gráfico a seguir mostra o tempo que o Spark e a abordagem Convencional levam para executar o processamento para tamanhos diferentes de entradas.
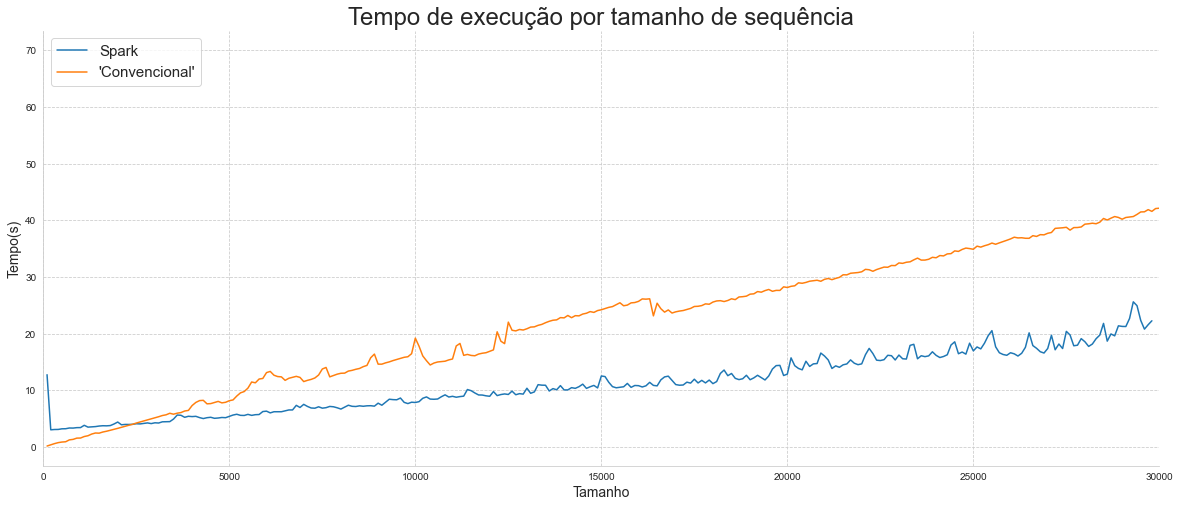
Na figura anterior podemos observar que o tempo utilizado pelo Spark é maior até um certo limiar, a partir desse ponto vemos que a utilização do Spark faz bastante diferença quando comparado ao algoritmo tradicional. Essa diferença ocorre devido ao tempo necessário para a criação das partições, vemos também que a primeira iteração do Spark leva mais tempo, porém, de maneira geral, conseguimos perceber que quanto maior o dado mais interessante se faz o uso do Spark. Para gerar o resultado acima criamos um algoritmo que gera sequências artificiais de diferentes tamanhos, executam a contagem e registram o tempo de maneira automática. Os códigos estão disponíveis no Github.
Glossário
Call-by-need: é uma teoria de linguagem de programação também conhecida como “lazy evaluation”, que traduzindo para o português significa “avaliação preguiçosa”, que consiste no atraso de execução de uma ação até o momento em que ela é considerada necessária. A utilização desta teoria permite um aumento no desempenho ao evitar ações desnecessárias, evitar erros nas avaliações e entre outros inúmeros benefícios.
Processamento in-memory: é uma aplicação da computação in-memory, que é quando os dados são alocados na memória principal do computador e não nos discos rígidos, como de costume, com isso a velocidade para análise é bem maior pois os dados já estarão disponíveis instantaneamente.
Hadoop Files System (HDFS): é um sistema de arquivos distribuído que lida com grandes conjuntos de dados executados em hardware comum. Ele é usado para dimensionar um único cluster Apache Hadoop para inúmeros nós.
SparkContext: é o objeto que permite a porta de entrada do Spark ao programa que está sendo desenvolvido. Ele pode ser acessado como uma variável em um programa para utilizar os seus recursos.
SNP: Polimorfismo de nucleotídeo único ou polimorfismo de nucleotídeo simples em genética, (em inglês, single nucleotide polymorphism; SNP) é uma variação na sequência de DNA que afeta somente uma base na sequência do genoma entre indivíduos de uma espécie.
Referências
DAMJI, J. S. et al. Learning Spark: lightning-fast data analytics. Traducao . [s.l.] O'Reilly Media, 2020.
Documentation: Apache Spark. Disponível em: https://spark.apache.org/documentation.html
GUO, R. et al. Bioinformatics applications on Apache Spark. Disponível em: https://academic.oup.com/gigascience/article/7/8/giy098/5067872?searchresult=1 Acesso em: 09 jul. 2021.
GUO, R. et al. Bioinformatics applications on Apache Spark. GigaScience, 2018.
Lazy Evaluation in Apache Spark – A Quick guide. Disponível em: https://data-flair.training/blogs/apache-spark-lazy-evaluation/ Acesso em: 09 jul. 2021
LEITE, M. O que é in-memory computing? Disponível em: https://www.artsoftsistemas.com.br/blog/o-que-e-in-memory-computing. Acesso em: 09 jul. 2021