por Renata Cavalcante

Legenda: Australian lungfish (Neoceratodus forsteri) no National Zoo & Aquarium em Canberra, Austrália. Créditos da imagem: Mitch Ames / CC BY-SA 4.0.
Qual animal com o maior genoma já sequenciado?
A resposta para esta pergunta é Neoceratodus forsteri, conhecido popularmente como peixe-pulmonado australiano (Australian lungfish), tornou-se o organismo animal com o maior genoma já sequenciado, sendo o recorde anterior do Ambystoma mexicanum, uma espécie de salamandra, com seu genoma de 32 Gbp. Com o estudo publicado em janeiro de 2021 na Nature o pesquisador Axel Meyer e os seus colaboradores mostraram que o genoma do Australian lungfish possui 43 bilhões de pares de bases (43 Gbp), o que é cerca de 14 vezes maior do que o genoma humano (2.9 Gbp).
Para auxiliar no desafio que foi a montagem deste grandioso genoma, Meyer e seus colaboradores utilizaram a tecnologia Nanopore long-read e ultra-long-read juntamente com um algoritmo de montagem - MARVEL assembler - desenvolvido anteriormente pelo grupo do pesquisador Siegfried Schloissnig, também colaborador do trabalho com o Australian lungfish. A fim de corrigir inserções e/ou deleções, gaps, polimorfismos de nucleotídeo único entre outros fatores foram utilizados dados de DNA e RNA oriundos de sequenciamento via Illumina. Outro método utilizado foi a captura de conformação cromossômica para auxiliar na montagem a nível de scaffold, para isto foi empregada a tecnologia Hi-C, que consiste basicamente em utilizar a conformação da cromatina para sequenciar porções de nucleotídeos que estão próximos no espaço 3D, mas podem estar separados por muitos pares de bases no genoma linear. Todos esses métodos contribuíram para a montagem do colossal genoma do Australian lungfish.
O vasto tamanho deste genoma é atribuível principalmente a presença de grandes regiões intergênicas e introns com alto teor de repetição. De acordo com o grupo, cerca de 90% do genoma do Australian lungfish corresponde a regiões repetitivas, cujos componentes assemelham-se aos de organismos tetrápodes (sendo composto principalmente por longos elementos nucleares intercalados).
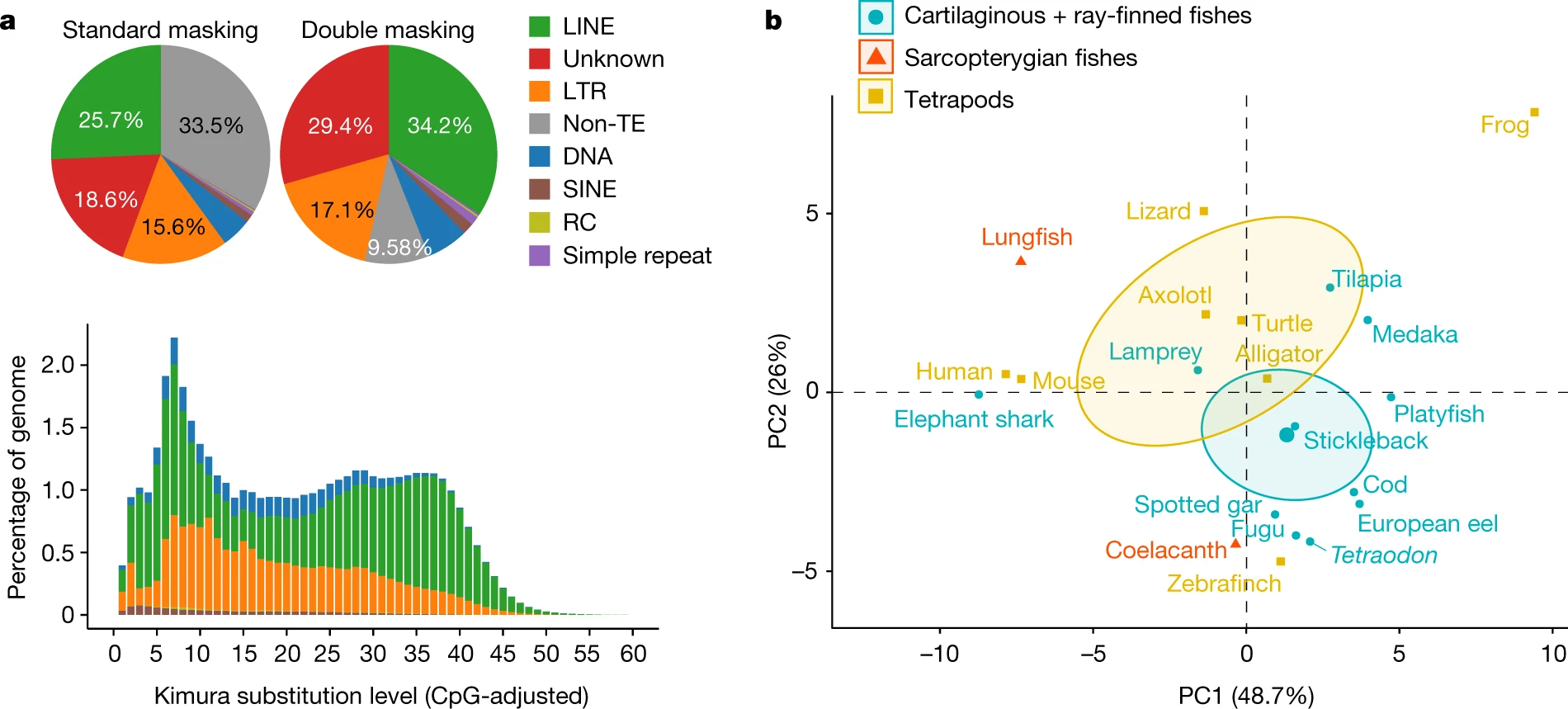
Legenda: Fig. a: Os gráficos de pizza mostram a composição geral de elementos repetitivos de conjunto não identificado (primeira anotação de elemento transponível) (esquerda), juntamente com a anotação do genoma mascarado (segunda anotação de elemento transponível) (direita). O gráfico de barras mostra a porcentagem no genoma das principais classes de elementos transponíveis. Nível de substituição Kimura (%) para cada cópia contra a sua sequência de consenso usado como proxy para o histórico de expansão dos elementos transponíveis. Cópias mais antigas (expansão antiga) acumularam mais mutações e mostram maior divergência das sequências de consenso. RC, transposões circulares; SINE, elemento nuclear intercalado curto; ET, elemento transponível. Fig. b: Componente principal (PC) análise da composição de elementos repetitivos (LTR, LINHA, SENO, DNA e desconhecido) de vertebrados. Fonte: https://www.nature.com/articles/s41586-021-03198-8/figures/3
Além do longo genoma de Australian lungfish, o estudo investigou o relacionamento deste grupo de peixes (peixes pulmonados) com o fluxo evolutivo que deu origem aos animais tetrápodes, de forma a tentar elucidar discussões anteriormente debatidas. Uma delas é relação de peixes-pulmonados ou celacantos - peixes arcaicos encontrados no Oceano Índico, conhecidos como “fósseis vivos” - com vertebrados terrestres.
A análise genômica deste estudo elucida que o peixe-pulmonado está mais intimamente ligado à linhagem evolutiva que deu origem aos tetrápodes, tendo então uma divergência mais inicial dentro o grupo dos celacantos, enquanto que os peixes pulmonados se ramificaram há 420 milhões de anos. O grupo olhou também para o número de genes associados com o desenvolvimento de pulmões e membros articulados, bem como a detecção de cheiros transmitidos pelo ar, e nesta análise foi visto que o Australian lungfish se assemelha ao grupo dos anfíbios quando se trata do número bruto destes genes.
Estas análises genômicas e filogenéticas endossam hipóteses anteriores de que os peixes pulmonados ocupam uma posição evolutiva chave, sendo o parente vivo mais próximo dos tetrápodes. No nosso país temos um representante de peixe pulmonado na Bacia Amazônica, conhecido popularmente por South American lungfish ou Pirambóia (Lepidosiren paradoxa), que ainda não possui genoma sequenciado. O que nos leva a refletir como seria interessante estudos com esse organismo vivente em nosso país para auxiliar na compreensão das inovações evolutivas associadas à terrestrialização das formas viventes no período Devoniano.
Referências:
Paper original genoma Neoceratodus forsteri, Nature:
Giant lungfish genome elucidates the conquest of land by vertebrates
Matéria NewScientist:
Australian lungfish has largest genome of any animal sequenced so far
Paper original genoma Ambystoma mexicanum, Nature:
The axolotl genome and the evolution of key tissue formation regulators
Paper original genoma humano, Science:
Paper original Hi-C, Science:
Comprehensive mapping of long range interactions reveals folding principles of the human genome