por Mayanne Aline Maia de Freitas

Se algum dia você se sentir triste, sem valor, que nada que você faz tem sentido, primeiramente você deve buscar ajuda profissional, e segundamente lembrar que para a microbiota que habita o seu corpo, você é o mundo inteirinho!
Você sabia que mais da metade do seu corpo não é humano? Apenas 43% das células que habitam em nós são células humanas, o restante é composta por microrganismos !
Com os avanços das tecnologias de sequenciamento e seu constante barateamento, temos cada vez mais acesso às informações escondidas no DNA. Há até pouco tempo atrás, estudos sobre comunidades microbianas associadas a algum organismo ou determinada superfície, por exemplo, possuíam bem menos poder informativo do que é possível ter hoje em dia. Porém, como dizia Tio Ben: “com grandes poderes, vem grandes responsabilidades”, e a preocupação hoje em dia, é saber analisar os dados gerados da maneira mais adequada possível.
A forma mais comum de analisar microbiomas é através do uso de genes marcadores de taxonomia (16S rRNA para bactérias e arqueias, ITS para fungos e 18S rRNA para os outros eucariotos), alguns bancos de dados e muita bioinformática! Algumas das ferramentas mais utilizadas hoje em dia são o Mothur e o Qiime 2, e apesar de existirem algumas diferenças “filosóficas” entre elas, como a linguagem de programação que para o Mothur são C and C++, e para o Qiime2 é o Python, podemos dizer que de forma bem simplificada, a maior parte das diferenças entre essas duas ferramentas são estéticas e pessoais (para uma discussão mais acalorada sobre o tema: (Qiime2 x Mothu). Por ora vamos falar um pouco sobre o Qiime2, e conhecer o que essa plataforma têm a nos oferecer.
O Qiime 2 (quantitative insight into microbial ecology) é um poderoso pacote especializado em análise de microbiomas que permite que o pesquisador inicie a análise com dados brutos de sequência de DNA e finalize com resultados estatísticos e figuras adequadas para publicação. O Qiime 2 têm como principais características ser descentralizado, extensível, ou seja, possui um sistema de plugins que estendem a sua funcionalidade, possui um sistema semântico que facilita a interpretação dos dados de entrada através da indicação do tipo de dados que estamos fornecendo, por exemplo: sequências emparelhadas com qualidade, matriz de distância, árvore filogenética, etc. A ferramenta também é capaz de suportar vários tipos de interfaces de usuário ( API, linha de comando, interface gráfica). Além disso, por ser focado na análise e transparência de dados, possui rastreamento integrado e automático da proveniência dos dados.
Quando os dados brutos (sequências de DNA) são importados para o Qiime 2, eles se transformam em um arquivo do tipo .qza (qiime zipped artifact) denominado artefato. Esses arquivos contêm os dados e algumas informações sobre o tipo de dado e a forma como este foi gerado. O uso de artefatos ao invés de arquivos de dados simples, como o FASTA, por exemplo, permite que os pesquisadores se concentrem somente na análise que deseja executar, sem se preocupar se os dados devem estar em um formato específico. Os artefatos podem ser utilizados como entrada para outras análises ou fornecerem informações simplesmente como estão, mas em boa parte das vezes, um artefato que serviu de entrada para uma determinada análise, gera como saída um arquivo do tipo .qzv (qiime zipped visualization), uma visualização. Arquivos do tipo .qzv não são usados como entrada para análises seguintes e sim para representar figuras, tabelas e resumos de dados estatísticos.
Além dos dois tipos de dados (artefatos e visualização), temos dois tipos de ações que podem ser executadas: método e visualizador. No método usamos artefatos e parâmetros como entrada, e como resultado podem ser gerados um ou mais artefatos como saída. Esses artefatos de saída podem subsequentemente ser usados como entrada para outros métodos ou visualizadores. O visualizador é semelhante ao método, pois também aceita uma combinação de artefatos e parâmetros como entrada, mas a diferença é que ele sempre gera uma visualização como saída.
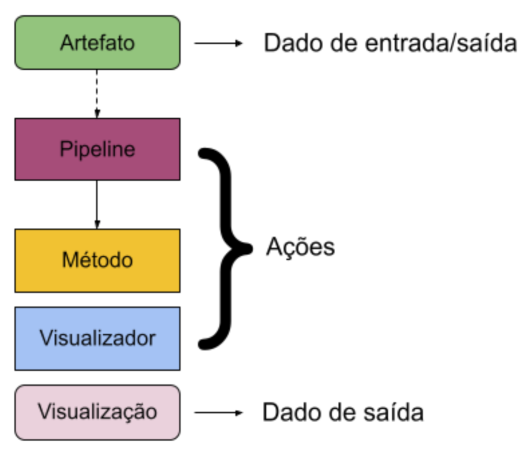
Figura 1. Fluxograma ilustrando os tipos de arquivos que são utilizados no Qiime 2, e as ações que podem ser executadas a partir dos mesmos.
O Qiime 2 pode ser instalado de forma nativa (recomendado) ou usando máquinas virtuais , e pode ser executado em todos os principais sistemas operacionais (Linux, macOS e Windows). Ele possui uma vasta documentação e mais recentemente conta com um canal no Youtube onde é possível acompanhar um workshop oferecido pelos próprios desenvolvedores da ferramenta.
Glossário:
API : A sigla API deriva da expressão inglesa Application Programming Interface que, traduzida para o português, pode ser compreendida como uma interface de programação de aplicação. Ou seja, API é um conjunto de normas que possibilita a comunicação entre plataformas através de uma série de padrões e protocolos.
Máquina virtual: A máquina virtual, ou VM, é um ambiente virtual que funciona como um sistema de computação com sua própria CPU, memória, interface de rede e armazenamento. Esse sistema virtual é criado a partir de um sistema de hardware físico.
Microbioma: a composição de todos os genes microbianos em uma comunidade, não confundir com microbiota que é um agregado de microrganismos, incluindo bactérias, arquéias, protistas, fungos e vírus
Pipeline: é um conjunto de elementos de processamento de dados conectados em série, onde a saída de um elemento é a entrada do próximo. Os pipelines têm como objetivo refinar e analisar os dados conforme necessário, o que resulta nas visualizações, análises e interpretações desejadas desses dados.
Referências
https://docs.qiime2.org/2021.4/about/
https://docs.qiime2.org/2021.4/concepts/
https://mothur.org/blog/2016/mothur-and-qiime/
https://docs.qiime2.org/2021.4/tutorials/overview/
https://www.bbc.com/portuguese/geral-43716220