por Maria Júlia Davi

Apenas analisando a palavra metagenômica, podemos inferir que esta área da ciência tem como objeto de estudo o conjunto (do “meta”: junto) de genomas. Mas de quem e de onde vem esse conjunto de genomas? O que é possível fazer com os dados gerados por essa ciência e qual a importância?
Os protagonistas da metagenômica são os microrganismos (bactérias, vírus, fungos) e como eles existem em praticamente qualquer ambiente, inclusive em seres humanos, animais e plantas, o conjunto de genomas a ser obtido depende do que se deseja descobrir e analisar. A aplicação inicial, e ainda a mais buscada até hoje, é a classificação taxonômica dos microrganismos presentes em um determinado ambiente.
Antes de ser possível classificar taxonomicamente os microrganismos por meio da metagenômica, as técnicas empregadas para este fim dependiam do cultivo dos mesmos em laboratório. As técnicas de cultivo apresentam diversas desvantagens: demandam muito tempo, são laboriosas, não contemplam microrganismos exigentes ou que não são possíveis de serem cultivados em laboratório e a identificação não se baseia em evidências moleculares, mas sim morfológicas e bioquímicas (o que pode levar a uma identificação incorreta).
Com o surgimento da metagenômica, o protocolo padrão se resume basicamente à extração do DNA de um ambiente, fragmentação, amplificação e sequenciamento deste material genético, que pode então ser analisado por ferramentas de bioinformática que fazem a organização taxonômica destas sequências. O tempo necessário para o processo inteiro depende da complexidade da amostra e plataforma de sequenciamento utilizada, mas em geral o tempo mínimo entre preparação da amostra para sequenciamento e obter o resultado final pode ser de 24 horas, segundo serviços ofertados por empresas de biotecnologia[1].
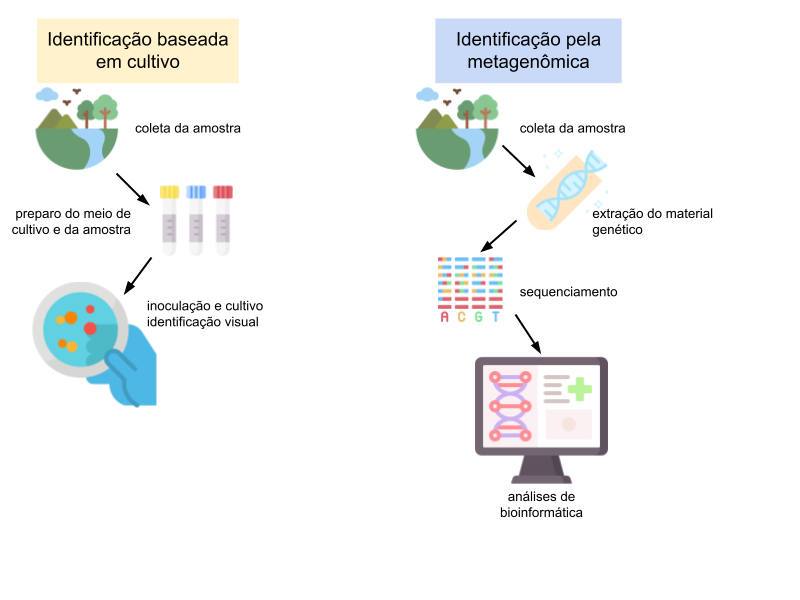 Legenda: Comparação resumida dos processos de identificação taxonômica pelo método tradicional de cultivo (esquerda) e pela metagenômica (direita).
Legenda: Comparação resumida dos processos de identificação taxonômica pelo método tradicional de cultivo (esquerda) e pela metagenômica (direita).
Com o objetivo de otimizar o processo de classificação, surgiram metodologias diferentes para obtenção das sequências. No sequenciamento shotgun, o DNA é extraído e passa por um processo de fragmentação e amplificação não-direcionada, o que significa que todo o conteúdo genético presente deve ser sequenciado. Já nas metodologias direcionadas, conhecidas como metagenômica de amplicons, são sequenciados apenas marcadores filogenéticos específicos para determinado grupo (exemplo: 16S para bactérias, ITS para fungos).
Com a metagenômica shotgun é possível realizar a identificação dos genes presentes nos dados gerados, o que leva a uma análise mais sofisticada. Exemplos incluem desde a identificação de genes de resistência a antibióticos, como também a identificação de genes que tenham funções metabólicas de interesse econômico ou ambiental.
Todo esse trabalho era e ainda é realizado, pois classificar taxonomicamente um ser vivo é importante para estabelecimento de protocolos como identificação de agentes patogênicos e diagnóstico de doenças, estudos de ecossistemas microbianos por meio da análise da diversidade de espécies, otimização de processos industriais, controle de qualidade na cadeia de produção de alimentos, entre outros.
Para que toda essa informação seja gerada, chegamos à questão de quais ferramentas de bioinformática utilizar para processar os dados. Existem algumas possibilidades: empresas que ofertam o serviço desde o sequenciamento até a análise dos dados; ferramentas pagas; ferramentas gratuitas. A escolha vai depender do tempo e recursos (financeiros e estruturais) disponíveis.
Dentre as empresas que disponibilizam o serviço completo, podemos citar a Illumina e Thermo Fisher, sendo a Illumina a empresa dominante em relação às plataformas de sequenciamento mais utilizadas atualmente. A Illumina também oferece a possibilidade de utilizar sua plataforma de análise de dados baseada na nuvem, por meio de um sistema de créditos, de acordo com a necessidade do usuário.
Algumas ferramentas pagas, como a Gaia, oferecem uma interface web amigável e possibilidade de realizar desde a análise taxonômica até a análise funcional da amostra, bem como a comparação de diferentes amostras com análises estatísticas. Outra plataforma web e gratuita, é o MG-RAST, que funciona como um servidor para análises metagenômicas.
No caso das ferramentas gratuitas, o usuário deve selecionar os programas necessários para realizar as etapas básicas do processamento das sequências e as etapas de classificação e visualização do resultado. O conjunto de programas selecionados e a ordem em que estes são executados para gerar o resultado que se deseja obter ao final do processo é definido como pipeline
Em relação aos programas gratuitos que realizam a etapa de classificação taxonômica do _pipeline_, podemos citar alguns desenvolvidos para o ambiente Linux e que podem ser executados via linha de comando: [KrakenUniq](https://genomebiology.biomedcentral.com/articles/10.1186/s13059-018-1568-0), [Clark](https://bmcgenomics.biomedcentral.com/articles/10.1186/s12864-015-1419-2), [GOTTCHA](https://pubmed.ncbi.nlm.nih.gov/25765641/), [Centrifuge](https://pubmed.ncbi.nlm.nih.gov/27852649/), [MetaPhIAn2](https://www.nature.com/articles/nmeth.3589), [Kaiju](https://www.nature.com/articles/ncomms11257) (também com versão web) e [MEGAN](https://www.ncbi.nlm.nih.gov/pmc/articles/PMC1800929/), este último disponível também em versão com interface gráfica e com funcionalidades adicionais além da classificação taxonômica.
Alguns desses programas podem ser executados em computadores comuns, que possuam a partir de 8 GB de memória RAM disponível, como é o caso do GOTTCHA, MEGAN e Centrifuge, e dependendo do banco de dados também o Kaiju. Esse é um diferencial que possibilita a execução do pipeline mesmo quando o usuário não possui um alto poder computacional disponível.
A implementação de ferramentas de análise cada vez mais eficientes e acessíveis é um avanço necessário, já que a metagenômica pode auxiliar no entendimento de muitas questões importantes e ultimamente também vem sendo bastante utilizada como metodologia principal em protocolos de monitoramento de patógenos emergentes. Abaixo uma tabela comparando alguns aspectos dos programas citados na matéria.
Tabela 1: Comparação entre as funcionalidades básicas dos programas mencionados.
| Programa | Funcionalidades | Gratuito |
| MG-RAST | Interface Web Análise funcional e taxonômica | Sim |
| KrakenUniq | Classificação taxonômica | Sim |
| Clark | Classificação taxonômica | Sim |
| GOTTCHA | Classificação taxonômica | Sim |
| Centrifuge | Classificação taxonômica | Sim |
| Kaiju | Interface Web Classificação taxonômica | Sim |
| MEGAN | Disponível para Windows, Linux e MacOS Analisa sequências classificadas por outros classificadores (DIAMOND, blast, etc) | Sim |
| Illumina BaseSpace | Interface Web Diversas análises, incluindo a classificação taxonômica | Não |
| Gaia | Interface Web Diversas análises, incluindo a classificação taxonômica | Não |
| MetaPhIAn2 | Classificação taxonômica por marcadores moleculares | Sim |
Glossário:
- 16S: termo que se refere ao gene da porção 16S do RNA ribossomal de procariotos, uma região bastante conservada e comumente utilizada para análises filogenéticas.
- Análise funcional: análise que busca identificar os genes presentes nas sequências disponíveis, possibilitando que as funções dos genes identificados sejam anotadas.
- Classificação taxonômica: sistema de classificação biológica que utiliza uma nomenclatura específica para denominar os diferentes níveis de organização dos seres vivos. Estes níveis correspondem a Domínio, Reino, Filo, Classe, Ordem, Família, Gênero e Espécie.
- ITS: sigla para Internal Transcribed Spacer, região não-funcional de RNA situada entre RNAs ribossomais estruturais de fungos, conservada entre as espécies e comumente utilizada para análises filogenéticas.
Referências
- Thermo Fisher Scientific. NGS Food Screening for Species Identification. Disponível em:<www.thermofisher.com/br/en/home/industrial/food-beverage/food-authenticity-labeling/ngs-food-screening-species-identification.html>
- Illumina BaseSpace Hub. Disponível em: <https://www.illumina.com/products/by-type/informatics-products/basespace-sequence-hub.html>
- GAIA: metagenomics data analysis software - sequentia biotech. Disponível em: <https://metagenomics.sequentiabiotech.com>
- MG-RAST: https://www.mg-rast.org
- KrakenUniq: KrakenUniq: confident and fast metagenomics classification using unique k-mer counts.
- CLARK: CLARK: fast and accurate classification of metagenomic and genomic sequences using discriminative k-mers.
- GOTTCHA: Accurate read-based metagenome characterization using a hierarchical suite of unique signatures.
- Centrifuge: Centrifuge: rapid and sensitive classification of metagenomic sequences
- MetaPhIAn2: MetaPhlAn2 for enhanced metagenomic taxonomic profiling
- Kaiju: Fast and sensitive taxonomic classification for metagenomics with Kaiju
- MEGAN: MEGAN analysis of metagenomic data