por Pitágoras Alves
RNAs longos não-codantes (lncRNAs) são um dos diversos tipos de RNA não-codante. Essas moléculas estão envolvidas em processos biológicos como regulação transcricional, interferência pós-transcricional e controle de tradução. Além disso, também há evidências de que estejam envolvidos no desenvolvimento de várias doenças humanas.
Até hoje, a identificação de lncRNA em muitas espécies diferentes permanece sendo um desafio, pois requer conhecimentos prévios como, por exemplo, anotações bem estabelecidas e dados de treinamento em espécies específicas. Infelizmente, poucas espécies têm esses dados disponíveis. Em um artigo recente de setembro na Bioinformatics, entitulado “Characterization and identification of long non-coding RNAs based on feature relationship”, pesquisadores apresentaram um novo software para tentar resolver esse problema: o LGC. O nome é uma sigla para “ORF Length and GC Content” e o objetivo é ser uma nova alternativa para a identificação de lncRNAs, de forma que os resultados não dependam de alinhamentos ou sejam enviesados para as espécie de onde vieram os dados de treinamento.
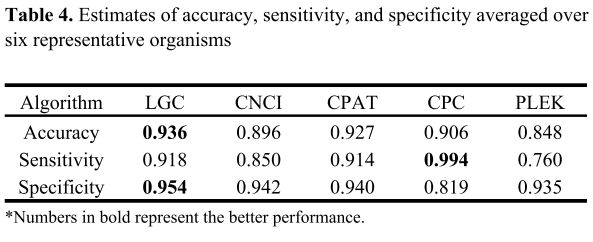
O principal diferencial desta ferramenta é que diferentemente das outras ferramentas, as quais analisam características da sequência como variáveis separadas e independentes, LGC analisa a relação biológica entre o comprimento de Open Reading Frame e o conteúdo GC para diferenciar RNAs codantes dos lncRNAs. O artigo faz uma comparação com as ferramentas CNCI, CPAT, CPC e PLEK em termos de acurácia, sensibilidade e especificidade. LGC apresentou a melhor acurácia e especificidade, enquanto CPC teve a melhor sensibilidade.
LGC tem uma interface web bastante simples para que os usuários possam enviar sequências no formato fasta, as quais são analisadas sem que o usuário precise instalar nenhum software. Além disso, a ferramenta também está disponível para download em forma de um script escrito em Python 2.7, caso o usuário deseje realizar a identificação de lncRNA em um volume maior de dados. Em um teste realizado utilizando como dados de entrada 250 mil sequências de cDNA de vertebrados (provenientes do banco de dados NR2), utilizando uma máquina com 8GB de RAM e um processador Intel Core i5-3337-U, o LGC demorou 71 segundos para concluir o processamento, um desempenho bastante satisfatório.
Referências:
Artigo original: Characterization and identification of long non-coding RNAs based on feature relationship, Wang et all. Disponível em https://doi.org/10.1093/bioinformatics/btz008.
Online em https://bigd.big.ac.cn/lgc/calculator.
Download em https://bigd.big.ac.cn/biocode/tools/BT000004.